Polymorphisme génétique et variation
PDF
Le polymorphisme génétique est l’existence d’états alternatifs de l’ADN, déterminant la variation des niveaux supérieurs d’intégration de l’organisme. Il existe différentes sortes de modifications du génome (mutations). Les plus étudiées sont les substitutions nucléotidiques dans les régions codantes et régulatrices.
1. Définition
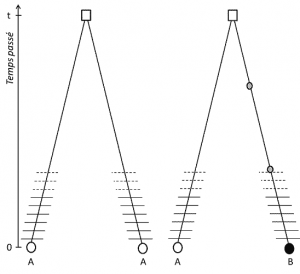
Le polymorphisme génétique est l’existence, dans une population, de plusieurs états alternatifs de l’ADN, ou allèles, en une position définie du génome, ou locusPosition du gène sur le chromosome. En génétique des populations, ensemble de gènes homologues (classe d’homologie). Deux gènes sont homologues s’ils s’apparient à la méiose. Pluriel : loci.. Cette définition a plusieurs aspects :
(1) D’abord le trait doit être porté par les chromosomes et être transmissible.
(2) Ensuite les allèles doivent être homologuesDeux chromosomes ou deux gènes sont dits homologues s’ils s’apparient et s’excluent mutuellement à la méiose. pour leur position dans le génome, ce qui exclut certains types de variations qui seront vus plus loin.
(3) Mais puisque le trait est transmissible, l’homologie de position implique aussi que les allèles soient homologues par descendance ; s’ils sont différents, c’est donc qu’une mutation (au moins) est intervenue dans l’une des lignées qui les relie à leur dernier ancêtre commun (figure 1).
(4) Enfin, le polymorphisme génétique peut être défini à l’échelle de la plus petite unité composant l’ADN : le site nucléotidique. De ce fait, chaque variant nucléotidique peut déterminer le polymorphisme des niveaux supérieurs de structuration biologique – le gène, la protéine et le phénotype de l’individu –, qui deviennent autant d’échelles auxquelles on peut parler de polymorphisme génétique (figure 2).
Prenons l’exemple du système sanguin ABO, qui correspond à des motifs antigéniquesPartie de l’antigène reconnue par un anticorps ou un récepteur lymphocytaire. Aussi nommé épitope ou déterminant antigénique. Un même antigène peut comporter plusieurs épitopes (identiques ou différents) et ainsi induire une réponse immunitaire variée. intervenant dans la compatibilité lors des transfusions sanguines. C’est un cas de polymorphisme génétique présent dans les populations humaines, portant sur la protéine ABO glycosyl-transféraseEnzyme permettant le transfert de résidus comportant des sucres sur des protéines. Dans le cas du système ABO, les glycosyl-transférase A et B induisent respectivement l’appartenance d’un individu au groupe A ou B. Lorsque les deux glycosyltransférases sont présentes, l’individu est de groupe AB. qui a trois allèles, A, B et O. Un individu peut être de génotype (AA), (AO) ; (BB), (BO) ; (OO) ou (AB). Il sera alors respectivement de phénotype [A], [A] ; [B], [B] ; [O] ou [AB], où l’on voit que A et B « dominent » O (c’est-à-dire que leur expression masque celle de O), et qu’ils sont co-dominants entre eux (l’hétérozygote entre A et B a une expression reconnaissable, [AB]).
Un polymorphisme peut être décrit premièrement à l’échelle de la séquence d’ADN du locus codant pour une protéine. Certains polymorphismes sont « synonymes », c’est-à-dire qu’ils ne changent pas la séquence d’acides aminés de la protéine ; ce sont souvent les polymorphismes les plus nombreux. Les autres modifient les acides aminés et sont dits polymorphismes de « remplacement ». Les polymorphismes de remplacement de l’ABO glycosyl-transférase sont de deux types : les polymorphismes changeant l’acide aminé mais pas le motif antigénique (ils n’interviennent donc pas dans le phénotype ABO des individus) ; et les autres polymorphismes de remplacement, qui déterminent le phénotypeEnsemble des caractères observables d’un individu. ABO.
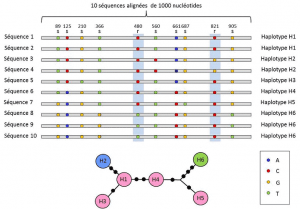
Entre le niveau du nucléotide et celui des allèles, les analyses génétiques tiennent compte d’un niveau de description intermédiaire : celui de l’arrangement linéaire des sites variables sur le locus. Ces arrangements, appelés haplotypesGroupe d’allèles de différents loci situés sur un même chromosome et habituellement transmis ensemble. Haplotype est un formé par la contraction de la locution anglaise haploid genotype, ou génotype haploïde. L’ensemble des gènes situés sur un même chromosome et dont les allèles ségrègent ensemble lors de la méiose constituent un haplotype. Ces gènes sont dits « génétiquement liés »., sont utiles en génétique évolutive car ils permettent de retrouver les liens généalogiques entre les allèles (figure 2).
Des polymorphismes nucléotidiques existent aussi dans les régions inter-géniques (hors des régions codantes). Certains affectent les zones de régulation de l’expression génétique et ont donc une expression phénotypique. D’autres n’ont pas d’effet connu et sont dits « silencieux ».
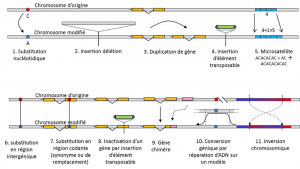
En plus des substitutions nucléotidiques, on parle de polymorphisme pour un type particulier de mutations, les microsatellites, qui correspondent à des variations de longueur d’un fragment d’ADN souvent non-codant (figure 3) dû au changement du nombre de répétitions dans une courte séquence répétée de nucléotides, par ex. CACACACA, ou TGTGTG.
Toute variation génétique n’entre pas dans la définition du polymorphisme génétique, puisque celle-ci exige l’homologie de position des alternatives (figure 3). Ainsi, il existe des séquences répétées obtenues par duplication d’un gène, pour lesquelles deux répétitions ne sont pas homologues par position. Souvent, on ne peut pas parler non plus d’homologie pour les éléments transposables, car ils changent généralement de position au cours des générations cellulaires, peuvent se multiplier et envahir le génome. L’homologie de deux éléments transposables ne peut alors être définie. Par contre, deux séquences codantes d’un même locus, dont l’une est intacte, et l’autre inactivée par l’insertion d’un élément transposable, correspondent bien à une situation d’homologie. On envisagera sans doute un jour de parler de polymorphisme pour les modifications des chromosomes dites « épigénétiques », qui sont parfois transmises sur plusieurs générations cellulaires, somatiquesQualifie les cellules non reproductrices, ou soma. Une mutation somatique, qui affecte un gène d’une cellule somatique disparaît avec l’individu porteur. ou germinalesQualifie les gamètes. Une mutation germinale peut être transmise à sa descendance., comme par exemple la méthylation. Leur importance dans l’évolution des populations reste à évaluer. La limitation de la notion de polymorphisme génétique à certaines catégories de variations – essentiellement les substitutions nucléotidiques – vient de la grande utilité de ces dernières pour explorer l’histoire des populations naturelles. Elles se prêtent à la modélisation mathématique de l’évolution.
Le mot de polymorphisme signifie « plusieurs formes ». Il s’oppose à monomorphisme, qui signale l’absence de variation. Dans le vocabulaire de la biologie, cette opposition a été longtemps utilisée, avant la génétique, pour signaler la coexistence de plusieurs types d’individus dans la même espèce, par ex. le polymorphisme de caste (reine et ouvrières) chez les insectes sociaux ; le polymorphisme saisonnier (changement de pelage) chez certains mammifères arctiques. Ces cas ne relèvent pas du polymorphisme génétique, concept plus récent ayant une signification plus étroite et plus précise. Les variations continues (par ex. la variation de taille) n’entrent pas non plus dans la catégorie du polymorphisme, car elles ne présentent pas d’alternatives distinctes. Par contre, les locus qui agissent sur la taille entrent dans cette définition. Nous parlerons ci-après des variations génétiques les plus étudiées car les plus importantes pour l’évolution, les substitutions nucléotidiques, puis nous parlerons de la variation des phénotypes.
2. Mesure
On ne peut se contenter de dire qu’un locus est plus ou moins variable sans donner à ce jugement une estimation quantitative. Un chercheur peut étudier la variation à différentes échelles. S’il s’intéresse seulement aux allèles d’une protéine, il mesurera la diversité allélique, symbolisée par ‘H’. S’il s’intéresse à la diversité de l’ADN, il mesurera la diversité nucléotidique, symbolisée par ’π’.
La diversité allélique H se définit comme la probabilité de tirer deux allèles différents lors de deux tirages avec remiseEffectuer un tirage successif avec remise de p jetons dans une urne qui en contient n, c’est prendre un premier jeton, relever sa valeur, le reposer dans l’urne, prendre un deuxième jeton, relever sa valeur, le reposer dans l’urne, etc. jusqu’au p-ième jeton. Cela revient à choisir p objets parmi n avec répétition (on peut choisir plusieurs fois le même objet) et avec ordre (l’ordre dans lequel on choisit les objets a de l’importance). Le nombre de tirages successifs avec remise de p jetons parmi n est : n × n × … × n = np.. Si l’on appelle pi la fréquence de l’allèle de rang i, on démontre que la probabilité de tirer deux fois le même allèle est F = Σ pi2. La diversité allélique de l’échantillon est alors son complément à 1, soit :
H = 1 – Σ pi2 (1)
Cette mesure s’applique tant aux allèles des protéines qu’aux haplotypes. On parle alors de diversité haplotypique. Elle peut aussi s’appeler hétérozygotie, car dans le cas d’un locus diploïde, elle donne la fréquence attendue des hétérozygotes.
La diversité nucléotidique π est l’équivalent de H à l’échelle de chaque nucléotide. On la calcule comme la moyenne du nombre de différences nucléotidiques entre les séquences d’un échantillon prises deux à deux (δij), divisée par la longueur du fragment d’ADN en nombre de nucléotides (L).
π = moyenne (δij)/L (2) (voir référence [1])
Cette valeur diffère selon les espèces. Dans les parties codantes du génome humain, deux chromosomes tirés au hasard dans la population diffèrent en moyenne à un nucléotide sur mille. Chez la mouche drosophile (Drosophila melanogaster) cette différence est d’environ un sur cent. La mouche est ainsi dix fois plus variable que l’homme. Quand ces valeurs sont rapportées à la taille des régions codantes (environ 15 500 gènes chez Drosophila, au moins 22 000 chez l’homme), voire à celle des génomes (140 millions de paires de bases par génome haploïde chez la mouche, environ 3 200 millions soit vingt fois plus chez l’homme), le nombre de sites polymorphes est astronomique et entraîne qu’aucun être né de la génération sexuée dans quelque espèce, n’est, n’a été et ne sera génétiquement identique à un autre être passé, présent ou à venir. La puissance d’information du polymorphisme de l’ADN est immense. En police scientifique, les détectives peuvent identifier n’importe quel suspect à partir de seize locus microsatellites.
3. Histoire
La variation est le titre des deux premiers chapitres du livre de Darwin l’Origine des espèces (1859). Darwin introduisit cette notion en sciences de la nature à une époque où les lois de l’hérédité biologique restaient un mystère, et lui consacra un autre livre important, la Variation chez les animaux et les plantes (1868). Convaincu que l’évolution portait sur de faibles variations modifiant peu l’adaptation des organismes à leurs conditions d’existence, il attribuait une importance cruciale aux petites variations quantitatives, ce qui incita ses successeurs (notamment Karl Pearson) à fonder la biométrieScience de la mesure du vivant. Désigne dans un sens très large l’étude quantitative des êtres vivants.. Mais la redécouverte des lois de Mendel en 1900 reporta l’intérêt sur les variations discontinues.
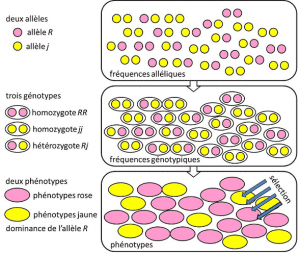
De 1908 à 1930, la génétique des populations fut essentiellement une discipline théorique cherchant à concilier le darwinisme avec l’hérédité mendélienneHérédité basée sur la transmission d’un seul gène sous un mode dominant, récessif ou lié au chromosome sexuel X (ou Y). Se dit d’un caractère génétique à déterminisme simple, par un couple ou un petit nombre de couples de gènes., et où les probabilités jouaient un rôle majeur. La génétique est contre-intuitive. Elle prédit qu’il n’y a pas de reproduction du parent dans son descendant, car le génotype de ce dernier résulte de la loterie de la séparation des allèles avant la fusion des deux demi-génomes parentaux. On réalisa que ce sont les fréquences alléliquesFréquence à laquelle se trouve l’allèle d’un variant dans une population. Exprimé comme une proportion ou un pourcentage. La somme des fréquences alléliques de tous les allèles d’un gène dans une population est donc par définition égale à 1. En génétique des populations, les fréquences alléliques représentent la diversité génétique au niveau de la population, ou de l’espèce. qui sont transmises entre générations, et non les génotypes ou les phénotypes. Ces fréquences sont à peu près stables d’une génération à l’autre et produisent, par-delà l’enchevêtrement multiple des filiations entre individus, les mêmes fréquences génotypiquesStructure génétique de la population. Déterminée à partir des fréquences alléliques. (figure 4). La distribution des génotypes de la population, associée à une variance, est donc seule prédictible. Vers 1930, les trois théoriciens Ronald Fisher, JBS Haldane et Sewall Wright contribuèrent à créer la notion de population mendéliennePopulation dont l’hérédité obéit aux lois de Mendel. [2]. Dans ce cadre, l’évolution associe de façon obligatoire trois niveaux structuraux : le gène, l’individu et la population. Cette association à trois peut être résumée par la formule suivante : l’évolution des populations est un changement de la fréquence des allèles (échelle du gène, mais aussi du groupe), changement dépendant du tri des phénotypes par la sélection (échelle des individus). La recherche empirique en génétique des populations se développa alors, mais fut limitée pendant longtemps par notre ignorance sur le fonctionnement des chromosomes (la structure de l’ADN fut comprise en 1953 et son séquençage commença en 1977), et dût se replier sur l’étude des quelques polymorphismes visibles, comme la coloration des élytres des coccinelles ou celle de la coquille des escargots (lire focus La controverse des escargots). De grands débats opposèrent les généticiens pour savoir si les populations naturelles étaient globalement polymorphes ou monomorphes, et si le polymorphisme était en soi avantageux ou non. L’étude de la variation à l’échelle des macromolécules dût attendre 1966 (pour les protéines) et 1983 (pour l’ADN) avant de répondre à ces questions.
4. Variations quantitatives et héritabilité
Quand nous regardons autour de nous, nous voyons des différences physiques entre les êtres, certaines complexes comme les traits du visage, d’autres faciles à mesurer comme le poids ou la taille. Le bon sens nous suggère qu’elles sont en partie transmissibles, bien que de façon difficile à préciser. Ces traits sont souvent polygéniquesSous la dépendance de nombreux gènes. On parle d’hérédité polygénique. Le diabète est une maladie polygénique., ce qui signifie qu’ils sont influencés par de nombreux locus. La génétique du développement, et son application à la comparaison des espèces (l’évo-dévo, pour « évolution du développement ») démêlent l’écheveau des interactions complexes qui font qu’au cours du développement les cellules du corps, toutes pourvues d’un bagage génétique identique, se différencient par épigenèse pour donner des tissus différents. On sait encore mal comment s’édifient les caractères complexes. Les phénotypes mesurables peuvent s’étudier par recours à la biométrie, basée sur les propriétés des distributions statistiques. Une mesure aux propriétés utiles est la varianceDans un échantillon d’individus chez lesquels on a mesuré un trait donné, la variance est la différence entre la moyenne du carré des valeurs et le carré de la moyenne des valeurs. Cette mesure, toujours positive, indique la dispersion des individus. [3]. Quand plusieurs causes indépendantes déterminent la variation d’un trait, leurs variances sont additives et leur somme donne la variance du trait. Si elles ne sont pas indépendantes, on ajoute à la somme des variances la somme des covariances. La variance totale Vt d’un trait phénotypique est telle que :
Vt = Vga+Vgd+Vgi+Ve
Vga, variance génétique additive, est la somme des variances dues à chaque locus indépendamment des autres ; Vgd, variance génétique de dominance, résulte de l’interaction des deux allèles d’un même locus ; Vgi, variance d’interaction entre locus, provient du fait que les locus d’un même individu interagissent ;Ve, variance de l’environnement, est ici supposée indépendante de la variance génétique.
La variance génétique de dominance est commune à deux enfants de mêmes parents, car ils partagent des effets de dominance dès lors qu’à un locus donné ils ont reçu les mêmes allèles de leurs deux parents, ce qui leur arrive à un locus sur quatre. Ils ne partagent pas ces effets avec leurs parents, donc ils se « ressemblent » plus entre eux qu’ils ne ressemblent à leurs parents, malgré le fait qu’ils partagent autant de gènes avec les uns et avec les autres. Certes, un parent peut, par hasard, avoir reçu les deux mêmes allèles qu’un de ses enfants à un locus donné. Ce hasard dépend de la fréquence des allèles dans la population. On voit que le degré de ressemblance d’un enfant avec son parent pour un trait donné est une propriété de la population.
Plus généralement, l’ensemble des composantes de la variance génétique (Vga+Vgd+Vgi) peut varier entre populations. Dans les lignées d’élevage, un cas fréquent est la consanguinité, qui épuise la variance génétique : de ce fait, un trait peut avoir la même moyenne dans deux populations, mais avoir une variation génétique différente. La capacité d’un éleveur à sélectionner un trait est donc une caractéristique de la lignée, et non du caractère.
La relation entre parents et enfant se mesure par l’héritabilité h2. Elle se définit comme le rapport de la variance génétique additive à la variance totale [4] :
h2 = Vga/Vt

(1) Corrélation de 1, où la taille des enfants est exactement celle des parents, sans différence de moyenne ni de variance. Les distributions marginales sont celle de la taille des parents moyens et des enfants. Les coordonnées des points (parents, enfants) sont situées sur la bissectrice des axes de coordonnées. Un parent-moyen de valeur a engendre un enfant de valeur a.
(2) Corrélation de 0, où la taille des enfants est indépendante de celle des parents. Un parent-moyen de valeur a engendre un enfant de valeur indifférente, au sein de la distribution des enfants. Les coordonnées des points sont situées sur un cercle ; l’espérance de leur valeur est située sur une droite parallèle à l’axe des parents (d).
(3) Corrélation parents-enfant intermédiaire entre 0 et 1. Les coordonnées des points sont situées sur une ellipse. L’espérance de leur valeur n’est plus située sur la bissectrice des axes de coordonnées (qui est aussi le grand axe de l’ellipse de la distribution), mais sur la droite de régression r. Un parent-moyen de valeur a engendre un enfant de valeur b (comprise entre 0 et a) avec une certaine variance. On note que le cas (3) est intermédiaire entre les cas (1) et (2).
Elle est figurée sous diverses hypothèses dans la figure 5. L’héritabilité permet de savoir si un trait peut être sélectionné. La figure 5-3 montre qu’un éleveur pourra, en une génération de croisement, déplacer la moyenne d’un caractère de la valeur 0 à la valeur b s’il sélectionne les individus de valeur a. On peut montrer que b = h2a. Pour sélectionner un trait, il est utile d’augmenter le rapport d’héritabilité en standardisant l’environnement (donc en diminuant la variance environnementale), et en se plaçant dans les conditions d’environnement qui favorisent l’apparition du trait. Le généticien Waddington a montré que, dans la nature, un changement d’environnement pouvait ainsi faire apparaître des traits nouveaux qui seront ensuite sélectionnés au cours de l’évolution.
Références et notes
Photo de couverture: Coquilles d’escargots Cepaea nemoralis & Cepaea hortensis. André Künzelmann, UFZ
[1] Cette formule peut s’écrire de façon plus conventionnelle comme: \pi =\frac{n(n-1))}{2L}\sum _{i=1}^{n-1}\sum_{j=i+1}^{n}\delta ij
[2] Fisher R.A. (1930) The Genetical Theory of Natural Selection, Clarendon Press, Oxford ; Haldane J.B.S. (1932) The causes of evolution. London: Harper & Brothers ; Wright S. Evolution and the Genetics of Populations. vols. 1, 2, 3, 4 ; New Edition. University of Chicago Press. 1984 (réédition par l’auteur de ses principaux résultats).
[3] Soit un échantillon d’individus chez lesquels on a mesuré le trait étudié, la variance peut se définir comme la différence entre la moyenne du carré des valeurs et le carré de la moyenne des valeurs. Cette mesure, toujours positive, indique la dispersion des individus.
[4] Il s’agit ici de l’héritabilité au sens strict. On appelle héritabilité au sens large le rapport de la somme des variances génétiques sur la variance totale. Cette formule donne l’héritabilité avec les deux parents (le « parent moyen ») ; l’estimation avec un seul parent donnerait h2/2.
L’Encyclopédie de l’environnement est publiée par l’Association des Encyclopédies de l’Environnement et de l’Énergie (www.a3e.fr), contractuellement liée à l’université Grenoble Alpes et à Grenoble INP, et parrainée par l’Académie des sciences.
Pour citer cet article : VEUILLE Michel (4 février 2019), Polymorphisme génétique et variation, Encyclopédie de l’Environnement. Consulté le 20 janvier 2025 [en ligne ISSN 2555-0950] url : https://www.encyclopedie-environnement.org/vivant/polymorphisme-genetique-variation/.
Les articles de l’Encyclopédie de l'environnement sont mis à disposition selon les termes de la licence Creative Commons BY-NC-SA qui autorise la reproduction sous réserve de : citer la source, ne pas en faire une utilisation commerciale, partager des conditions initiales à l’identique, reproduire à chaque réutilisation ou distribution la mention de cette licence Creative Commons BY-NC-SA.







